CRISPR-Cas9、TALEN、ZFN―遺伝子編集における競争
革新的な生物学的研究を行うためには、技術の進歩が重要です。ZFN、TALEN、CRISPR/CRISPR-Casはゲノム編集に革命を起こしました。
DNAゲノム編集について
ゲノム編集は、特定のゲノム領域におけるDNA配列の半永久的な改変であると解釈されます。以前は、長い相同アームを持つDNAテンプレートを目的のゲノム領域に送達することで、ゲノムの編集は実施されていました。このDNA配列がゲノムに挿入される仕組みは、相同組換えプロセスに基づいており、宿主のヌクレアーゼとDNA修復機構が相同DNAテンプレートを使用してゲノムを修復することによって生じます。修復に用いられるDNAテンプレートには選択マーカーも含まれており、抗生物質を用いた選択によって、目的の変異が挿入されたゲノム改変細胞を選別します。
このプロセスは手間がかかり、長鎖DNAテンプレートを合成し、送達しなければいけません。この手法は、多くの哺乳類の細胞種において非効率的であり、選択後にゲノムに挿入された選択カセットの除去等の複数の操作を行う必要がありました。現在では、ZFN、TALEN、CRISPR-Cas9の3種類の異なる人工ヌクレアーゼを利用できます。
ゲノム編集にヌクレアーゼを使用する時代
ゲノム工学の著しい躍進によって人工的にヌクレアーゼを構築できるようになり、標的DNA配列を特異的に認識し、切断することが可能になりました(表1)。
―最初に開発された人工エンドヌクレアーゼは、ZFN(zinc finger nuclease:ジンクフィンガーヌクレアーゼ)です。ZFNは、自然界に存在する転写因子ファミリーであるジンクフィンガータンパク質を基にしており、エンドヌクレアーゼであるFokIが融合されています1。
ジンクフィンガードメインは、3ヌクレオチド(3塩基)のDNA配列を認識します。したがって、ジンクフィンガードメインを複数連結すると、より長いDNA配列を認識し、標的に対する特異性を高めることができます。しかし、1列に連結したジンクフィンガーモチーフ(ジンクフィンガーアレイ)は、アレイ内で隣同士のジンクフィンガーがそれぞれの特異性に影響する場合があるため、ジンクフィンガーアレイの設計と配列の選択はとても困難な作業となり、多くの時間を必要とします。また、最終的な配列の特異性を予測するのは困難です。FokIエンドヌクレアーゼは、二量体として機能します。それは、2つのZFNがDNAの対向鎖に結合した時にのみ、二本鎖DNAの切断が生じることを意味します(図1A)。この機構は、標的配列内の近傍に存在する異なるヌクレオチド配列をそれぞれ認識するように設計された2つのZFNに基づいており、両方のZFNが同時に結合領域を認識して結合する必要があるため、オフターゲット効果を抑制することが可能です。
―TALEN(Transcription activator-like effector nuclease:転写活性因子様エフェクターヌクレアーゼ)は、細菌のTALEタンパク質とFokIヌクレアーゼからなる融合タンパク質です2。
ZFNと同様に、TALENの特異性はタンパク質‐DNA間の特異的結合に由来します。TALENの場合、単一のTALEは1つのヌクレオチド(1塩基)を認識し、TALEアレイはより長い配列と結合することができます(図1B)。各TALEの作用は、1ヌクレオチドのみに限定され、隣接するTALEの結合特異性に影響を及ぼさないため、TALENの構築はZFNと比較してはるかに容易です。ZFNと同様に、TALEモチーフは、FokIエンドヌクレアーゼと結合しており、DNAを切断する場合は二量体を形成する必要があります。つまり、標的DNA領域近傍の対向鎖に別のTALENが結合しなければなりません。
―CRISPR-Cas9(clustered regular interspaced short palindromic repeats-Cas9:クリスパーキャス9)システムは、細菌の免疫システムをベースとしています。
CRISPR-Cas9は、Cas9ヌクレアーゼと、tracrRNA(トランス活性化型crRNA)およびsgRNA(一本鎖ガイドRNA)の2種類のRNAで構成されており、有名なワトソン-クリック型塩基対の結合作用によって標的配列を認識します。この複合体が働くためには、PAM(protospacer adjacent motif)と呼ばれるDNAモチーフがゲノム上に存在しなければなりません。各Cas9タンパク質は、特定のPAM配列を認識します3,4(例:一般的に使用されるCas9は5’-NGG-3’を認識します)。DNAの切断は、Cas9ヌクレアーゼが行い、野生型Cas9の場合は二本鎖切断を、ニッカーゼと呼ばれるCas9変異体の場合は一本鎖切断を生じさせます(図1C)。CRISPR-Cas9機構におけるDNA領域の認識は、RNA-DNA相互作用によって制御されます。塩基配列による制御機構は、ZFNやTALENと比較して、ゲノムターゲットの構築やオフターゲット領域の予測が容易で、複数のゲノム領域を同時に改変できる可能性がある等、多くの利点を備えています。
| 特徴 | ZFN | TALEN | CRISPR-Cas9 |
| 認識する標的DNAの長さ | 9~18bp | 30~40bp | 22bp +PAM配列 |
| 標的DNAを認識するメカニズム | DNA-タンパク質相互作用 | DNA-タンパク質相互作用 | ワトソン-クリック型塩基対のDNA-RNA相互作用 |
| DNAを切断・修復するメカニズム | FokIによる二本鎖切断 | FokIによる二本鎖切断 | Cas9による一本鎖切断または二本鎖切断 |
| 設計 | 容易ではない。すでに標的への特異性が判明しているジンクフィンガーモチーフのライブラリが存在するが、一列に連結した場合、隣接するジンクフィンガーモチーフの特異性に影響を及ぼす可能性があるため、設計は容易ではない。 | 簡単。標的への特異性を有するTALEモチーフが多く判明している。 | 簡単。標的DNAと相補的なSgRNAをデザインする。 |
| クローニング | ジンクフィンガーモチーフ同士を遺伝子工学的に結合する必要がある。 |
TALEは、煩雑な結合操作を必要としない。別々のTALEモチーフのクローニングは、ゴールデンゲートアセンブリを使用して行うことができる5。 |
Cas9の発現ベクターを利用可能。SgRNAは、DNA発現ベクターとして細胞に送達するか、直接的にRNA分子を細胞に送達するか、事前にCas9-RNA複合体を調製して細胞に送達する。 |
表1. 一般的に使用されている人工ヌクレアーゼの標的特異性、作用機序、実験デザイン。
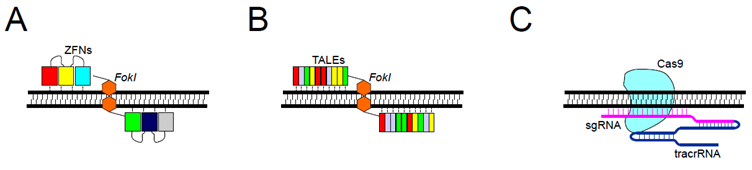
図1. 人工ヌクレアーゼ。A. ZFN―2つの個別のZFNが、向かい合うDNA鎖の特異的領域を認識して結合し、形成されたFokI二量体が標的DNAを特異的に切断する。B. TALEN―2つの個別のTALENが、向かい合うDNA鎖の特異的領域を認識して結合し、形成されたFokI二量体が標的DNAを特異的に切断する。C. CRISPR-Cas9機構では、標的DNA領域は、ゲノムDNAとsgRNAの塩基相補性によって認識される。このsgRNAにはtracrRNAが後に続き、これらのRNAと結合しているCas9ヌクレアーゼが標的DNA領域を取り込み、DNA切断を行う。
オフターゲット効果の低減
オフターゲット効果を低減する人工ヌクレアーゼの設計は、基礎科学的なアプローチの課題であるだけでなく、臨床で使用する場合や産業的な利用を想定した場合にも大きな課題です。
In vivoとin vitroのいずれも、目的部位へのZFNやTALENの送達は、オフターゲット領域への結合や不必要なDNA切断の誘導を原因とする、毒性や致死性を示す可能性があります。ZFNとTALENで使用されるFokIの変異は、2つの個別のヌクレアーゼが標的部位に結合し、二量体化している時にのみFokIエンドヌクレアーゼ活性を促進できるように導入されました6,7。CRISPR-Cas9機構のオフターゲット切断は、ほとんどの場合、sgRNAが完全に相補的なゲノム領域、または部分的に相補的なゲノム領域を認識することによって生じます8。選択的な送達手法やCas9の半減期の改変により、細胞内でCas9タンパク質が活性型になっている量と時間を限定するといった、オフターゲットによる切断を制限する様々なアプローチが提案されています9。オフターゲット特異性の低い、HF-Cas9、eCas9、HypaCas9等の、種々のCas変異体が作り出されています10-12。CRISPR-Cas12(Cpf1)やCRISPR-Cas13a(C2c2)のような、新たなCas9変異体やCas9ホモログは、異なるPAMを認識することができるため、ゲノム編集の正確性を大幅に向上させるだけでなく、高い標的特異性を示す可能性があります13。興味深い新案であるCas9とFokIヌクレアーゼの融合体は、ZFN/TALENとCRISPR機構の長所を兼ね備えています13,14。
新しい研究ツールとしてのCas9キメラ―DNA切断に留まらない使い方
機能欠失研究(loss-of-function)や機能獲得研究(gain-of-function)等の一環として、様々なCasキメラ体が作り出されています15。Cas9によってゲノムターゲティングが可能になると同時に、付加タンパク質融合体ではCas9活性の調節が可能になります。
「CRISPR干渉」(CRISPRi)では、KRABエフェクター抑制ドメインと融合したCas9タンパク質が遺伝子転写抑制によって遺伝子発現をダウンレギュレーションします16。このアプローチは、一般的なRNA干渉(RNAi)の手法よりも優れている点があり、タンパク質をコードしない遺伝子の機能を調べることができます。さらに、逆のアプローチとして「CRISPR活性化」(CRISPRa)も可能です。転写活性化ドメイン(VP64等)とCas9の融合体により、目的のゲノム上の遺伝子座の発現を増強します。別のタイプの転写活性化はSAM(synergistic activation mediator)の存在下で行われ、その際のCas9融合体には活性化効率を最大化するための複数の活性化ドメインが組み込まれています17。Cas9キメラの中には、ヌクレアーゼ活性を持たないものもあります。例えば、クロマチン修飾酵素とCas9の融合体はエピゲノム研究に使用され、タンパク質‐DNA結合部位の可視化、クロマチン構造の探索、ヒストンの翻訳後修飾の調査研究等が行われています18,19。
CRISPR-Cas9と細胞株の遺伝子編集のイノベーション
CRISPR-Cas9は、ヒトゲノムの有害な遺伝子変異を修復するために利用することができます。このアプローチでは、Cas9タンパク質、tracrRNA、領域特異的sgRNAと共に野生型配列のDNA修復テンプレートが必要となります。Cas9により部位特異的なDNA切断が生じ、生じた切断は続いて宿主のDNA修復機構により修復され、野生型アレルが復元します(図2A)。
CRISPR-Cas9は、細胞工学においても信頼できるツールです。このアプローチでは、Cas9タンパク質、tracrRNA、領域特異的sgRNAを送達する必要があります。Cas9は部位特異的にDNAを切断し、それは宿主の修復機構によって修復されます。修復用テンプレートが存在しない場合は、ヌル変異となるトランケーション(遺伝子の切り詰め・短縮化)が生じます。目的の変異配列を含む修復テンプレートを細胞へ導入することにより、よりターゲットを絞ったアプローチが可能です(図2B)。
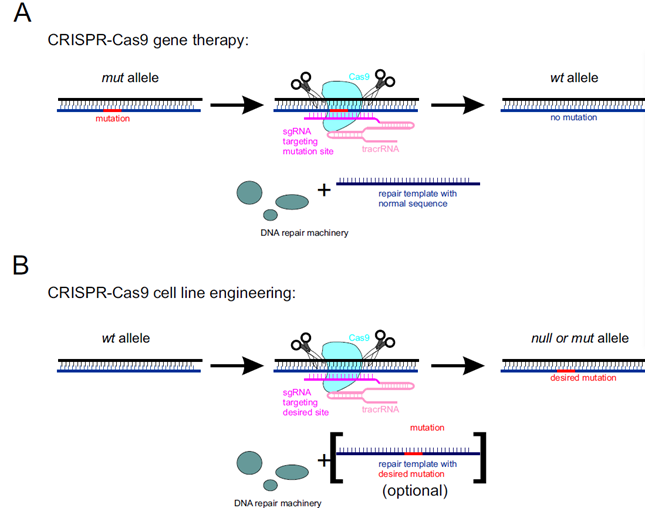
図2. A)遺伝子治療およびB)細胞工学におけるCRISPR-Cas9機構の利用。
今後の展開
人工ヌクレアーゼは、ゲノム工学に革命を起こし、哺乳類ゲノムの編集を容易にしました。ゲノム編集技術は、遺伝性疾患に対して、DNAレベルで原因遺伝子の変異を修復する可能性を持つメカニズムであるため、基礎研究だけではなく前臨床研究や臨床研究でも一般的に使用される可能性があります。基礎研究と応用研究における技術革新によって、わずか20年前に始まった技術が発展し続けた結果、将来的には先端科学や生物医学の分野で幅広く使用されると考えられます。
投稿者:Karolina Szczesna博士、プロテインテック シニアプロダクトマネージャー兼テクニカルサポート
参考文献
- Kim, Y. G., Cha, J. & Chandrasegaran, S. Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain. Proc. Natl. Acad. Sci. U. S. A. 93, 1156–1160 (1996).
- Christian, M. et al. Targeting DNA double-strand breaks with TAL effector nucleases. Genetics 186, 757–761 (2010).
- Mali, P. et al. RNA-guided human genome engineering via Cas9. Science 339, 823–826 (2013).
- Cong, L. et al. Multiplex genome engineering using CRISPR/Cas systems. Science 339, 819–823 (2013).
- Cermak, T. et al. Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting. Nucleic Acids Res. 39, e82 (2011).
- Szczepek, M. et al. Structure-based redesign of the dimerization interface reduces the toxicity of zinc-finger nucleases. Nat. Biotechnol. 25, 786–793 (2007).
- Ramalingam, S., Kandavelou, K., Rajenderan, R. & Chandrasegaran, S. Creating designed zinc-finger nucleases with minimal cytotoxicity. J. Mol. Biol. 405, 630–641 (2011).
- Zhang, X.-H., Tee, L. Y., Wang, X.-G., Huang, Q.-S. & Yang, S.-H. Off-target Effects in CRISPR/Cas9-mediated Genome Engineering. Mol. Ther. Nucleic Acids 4, e264 (2015).
- Maji, B. et al. Multidimensional chemical control of CRISPR-Cas9. Nat. Chem. Biol. 13, 9–11 (2017).
- Chen, J. S. et al. Enhanced proofreading governs CRISPR-Cas9 targeting accuracy. Nature 550, 407–410 (2017).
- Kleinstiver, B. P. et al. High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide off-target effects. Nature 529, 490–495 (2016).
- Slaymaker, I. M. et al. Rationally engineered Cas9 nucleases with improved specificity. Science 351, 84–88 (2016).
- Murugan, K., Babu, K., Sundaresan, R., Rajan, R. & Sashital, D. G. The Revolution Continues: Newly Discovered Systems Expand the CRISPR-Cas Toolkit. Mol. Cell 68, 15–25 (2017).
- Tsai, S. Q. et al. Dimeric CRISPR RNA-guided FokI nucleases for highly specific genome editing. Nat. Biotechnol. 32, 569–576 (2014).
- Ribeiro, L. F., Ribeiro, L. F. C., Barreto, M. Q. & Ward, R. J. Protein Engineering Strategies to Expand CRISPR-Cas9 Applications. Int. J. Genomics 2018, 1652567 (2018).
- Gilbert, L. A. et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell 154, 442–451 (2013).
- Konermann, S. et al. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. Nature 517, 583–588 (2015).
- Liu, X. S. et al. Editing DNA Methylation in the Mammalian Genome. Cell 167, 233-247.e17 (2016).
- Vasquez, J.-J., Wedel, C., Cosentino, R. O. & Siegel, T. N. Exploiting CRISPR-Cas9 technology to investigate individual histone modifications. Nucleic Acids Res. 46, e106 (2018).


