RIP/RIP-seqによるRNA修飾の研究
Sripriya Raja著(ピッツバーグ大学、PhD Candidate)
RIP/RIP-Seqとは?
RIP/RIP-seq(RIP:RNA immunoprecipitation、RIP-seq:RNA immunoprecipitation sequencing)は、RNAとRNA結合タンパク質との間の相互作用を研究するために用いられるハイスループットな実験技術です。RNA結合タンパク質に対する抗体を用いて免疫沈降を実施後、結合していたRNA分子を回収し、次世代シーケンス技術等を用いて解析を行います。RIP/RIP-Seqによって、細胞や組織内の特定のRNA結合タンパク質とターゲットRNA配列を同定することが可能になります。
RNA修飾を研究するうえでRIP/RIP-seqが有益な理由とは?
RNA修飾の研究は、RNA生物学の複雑性や遺伝子発現制御の理解、疾患メカニズムの解明、診断や治療への応用を試みるにあたり非常に重要です。RNA修飾を研究することで、細胞内プロセスや疾患におけるRNA分子とその役割をより深いレベルで理解することができます。
RNA修飾の調節不全は、がん、神経疾患、代謝疾患等の様々な疾患に関係があることが示唆されています。そのためRNA修飾の解明によるRNA修飾を標的とする治療法の開発が期待されています。例えば、特定のRNA修飾を付加または除去する作用を有する酵素を操作することで、疾患発症時における遺伝子発現の調節、RNA構造の調整、異常なRNA修飾の修正を実現できる可能性があります。さらに、RNA修飾の薬理的調節が可能になれば、治療介入の新たな道筋を開くことにもつながります。RNA修飾の概要とRNA修飾による機能的影響の解明は、RNAをターゲットとした治療法やPrecision medicine(精密医療/個別化医療)の開発に寄与する可能性があります。
RIP/RIP-seqは、RNA結合タンパク質のターゲット配列、RNA生物学におけるRNA結合タンパク質の機構的役割、遺伝子発現制御、疾患に関する重要な洞察をもたらします。RIP-seqにより、研究者はRNA結合タンパク質のターゲット配列に加えて、RNAスプライシング、RNAの安定性、輸送、翻訳といった様々な細胞内プロセスにおけるRNA結合タンパク質の制御的役割に関する識見を得ることができます。研究者は、異なる実験条件または異なる種類の細胞のRIP-seqデータを比較することで、RNA結合タンパク質の結合パターンの変化を調べ、遺伝子発現に隠された制御メカニズムを明らかにできる可能性があります。RIP-seqは、RNA‐タンパク質間相互作用の解明に寄与し、様々な生物学的プロセスや疾患におけるRNA結合タンパク質の役割を明らかにするうえで特に有用です。
RIP/RIP-seqの実施方法
RIPとは、RNA結合タンパク質とRNA分子を共に免疫沈降する手法です。RIP技術は、通例はホルムアルデヒドのような化学的架橋剤を使用してRNA結合タンパク質をRNAに架橋し、特定の時点におけるRNA結合タンパク質とRNA間の相互作用を「フリーズ(停止)」させることで実施されます。次に、架橋処理した細胞または組織のライセートを、目的のRNA結合タンパク質に特異的な抗体を使用した免疫沈降に供します。続いて、免役沈降で回収したRNA‐タンパク質複合体を精製し、脱架橋させることでRNA分子を溶出します。最後に濃縮RNAを解析し、RNA結合タンパク質が結合していた転写産物を同定します。
RIP-seqは、RIP技術にハイスループットなシーケンス技術を組み合わせた技術であり、RNA結合タンパク質が結合するRNA分子のゲノムワイドな同定および特性解析を可能にします。免疫沈降のステップの後に、精製RNAからcDNAライブラリーを作製し、次世代シーケンサーを使用してその配列を解析します。RIP-seqのデータからは、RNA結合タンパク質に結合していたRNAの配列に関する情報が得られ、研究対象とするRNA結合タンパク質がターゲットとする転写産物を同定することができます。

図1. RIP/RIP-seqのワークフロー
RIP/RIP-seqワークフローの概要
-
架橋:RIP-seqの第一のステップは、細胞内のRNA結合タンパク質とRNA分子の架橋です。通例、細胞をホルムアルデヒド等の化学架橋剤で処理することで架橋します。架橋によってRNA結合タンパク質‐RNA間の相互作用がそのままの状態で保存され、後続のステップの間にRNA結合タンパク質とRNA分子が解離するのを防ぎます。
-
細胞溶解および免疫沈降*1:架橋処理した細胞を溶解し、細胞内容物を放出させることで、免疫沈降用のライセートを調製します。ライセート溶液に目的のRNA結合タンパク質に特異的な抗体を添加すると、抗体は目的のRNA結合タンパク質に対し選択的に結合します。続いて、Protein A/Gビーズのような抗体に結合する担体を使用する手法によって、RNA結合タンパク質‐RNA複合体を免疫沈降します。
-
RNAの断片化:免疫沈降で回収したRNA‐タンパク質複合体をRNaseで処理し、RNA結合タンパク質との複合体を形成していないRNA分子/RNA領域を分解・消化することで、RNA結合タンパク質と結合したRNAのみを残します。続いて、RNA結合タンパク質と結合しているRNA分子が残るように、複合体からRNA結合タンパク質を除去します。
-
RNA精製:RNAは、タンパク質フラグメント、コンタミネーション物質、架橋物が残存する溶液から精製されます。このステップでは、高品質なRNAを回収するために様々な酵素処理やカラム精製等を実施します。
-
ライブラリー調製および配列決定:精製RNAはcDNAに逆転写され、アダプター配列を付加した配列決定用のライブラリーが調製されます。続いて、次世代シーケンシング(NGS)のようなプラットフォームを使用して、ライブラリーをハイスループットシーケンシングにかけ配列を読み取ります。
-
データ解析:得られたシーケンスリードを、リファレンスゲノムまたはトランスクリプトームにアライメントし、RNA結合タンパク質が結合していた領域に該当するRNA分子を特定します。データ解析では、RNA結合タンパク質の結合部位が集中して存在する領域(ピーク)の特定を行い、ダウンストリーム解析を実施し、RNA結合タンパク質‐RNA間相互作用の機能的影響を解明します。
*1:RNA結合タンパク質に対する抗体の代わりに、RNA修飾(トランスクリプトーム修飾)を認識する抗体を用いて免疫沈降(IP)を実施する場合もあります(例. RNAメチル化修飾の1つであるN6-メチルアデノシン(m6A)を標的とする抗体を用いたIP)。IP後、RNA結合タンパク質の検出(図2、図3)や、回収されたRNA領域の解析によるトランスクリプトーム全体におけるRNA修飾のマッピングが可能になります。
プロテインテックのRNAメチル化修飾研究用抗体を用いて免疫沈降を実施後、RNA結合タンパク質をウェスタンブロットで検出しました
m6Aモノクローナル抗体(カタログ番号:68055-1-lg)
 |
 |
|
図2. HEK-293細胞ライセートを免疫沈降し、YTHDF1抗体(カタログ番号:17479-1-AP)を使用して実施したウェスタンブロット。左:Protein A-マウスIgG3コントロール抗体を用いて実施した免疫沈降サンプル。中:Protein A- m6A抗体(カタログ番号:68055-1-Ig)を用いて実施した免疫沈降サンプル。右:Inputサンプル。 |
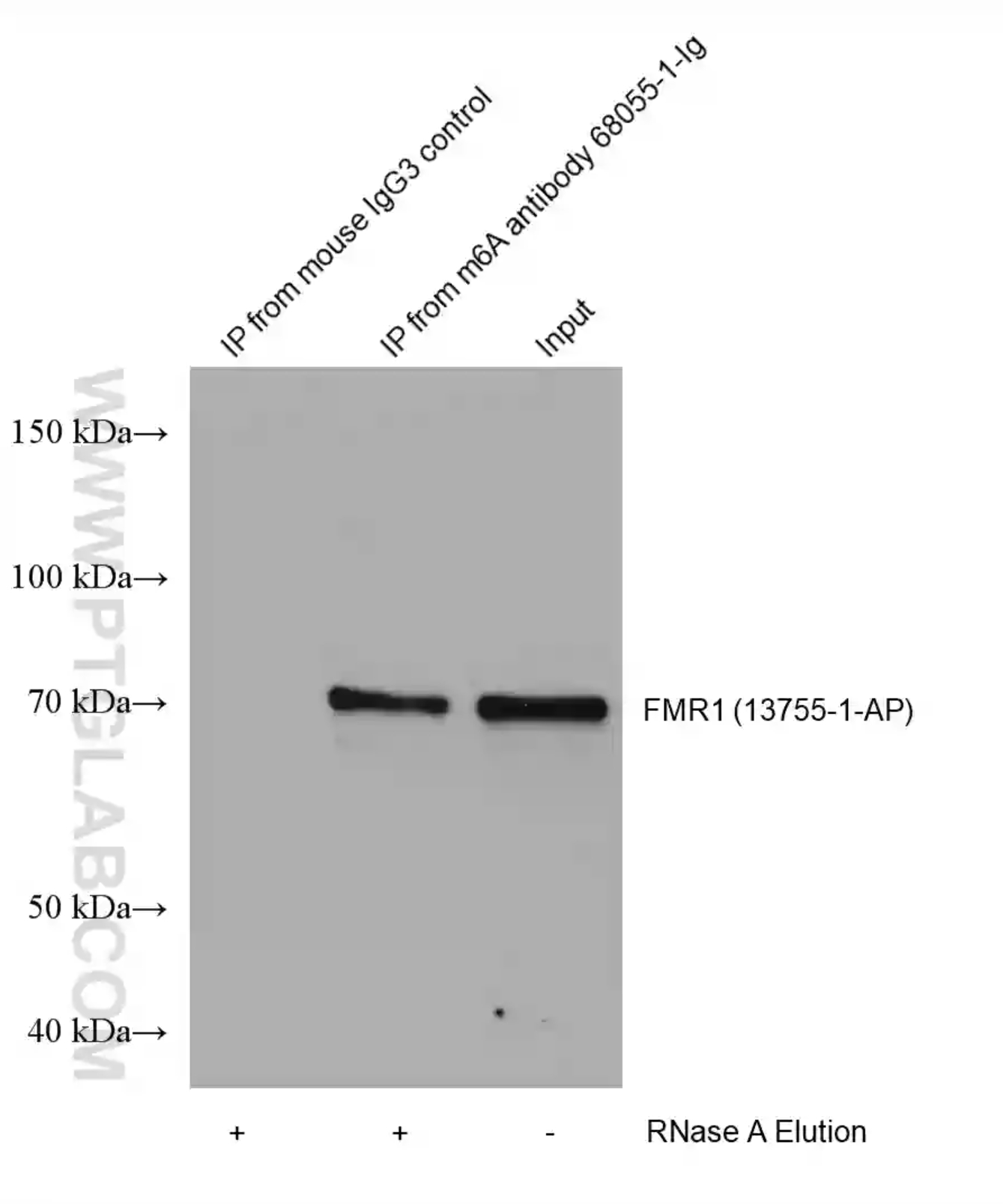
図3. HEK-293細胞ライセートを免疫沈降し、FMR1抗体(カタログ番号:13755-1-AP)を使用して実施したウェスタンブロット。左:Protein A-マウスIgG3コントロール抗体を用いて実施した免疫沈降サンプル。中:Protein A- m6A抗体(カタログ番号:68055-1-Ig)を用いて実施した免疫沈降サンプル。右:Inputサンプル。 |


