翻訳後修飾(PTM:Post Translational Modification)とは?
DNAの塩基配列に従って転写・翻訳されたタンパク質は、アミノ酸配列が同一であっても「翻訳後修飾」を受けることにより特性/機能が変化します。
|
目次 |
翻訳後修飾の概要
生物を構成する細胞は、内部環境や外部環境の状態の変化を感知し迅速に応答します。細胞を構成する重要な主成分の1つとしてタンパク質が存在し、タンパク質はDNAから転写されたmRNAに基づき「翻訳(translation)」と呼ばれる過程により生合成されます。生合成後のタンパク質は様々な化学修飾を受けることが知られており、翻訳されたタンパク質が受ける化学修飾は「翻訳後修飾(PTM:Post Translational Modification)」と呼ばれます。翻訳後修飾は細胞の応答を調節する仕組みの1つです。細胞内/細胞外の状態変化は、可逆的な翻訳後修飾を介してセンサー分子からエフェクター分子へと伝達されます。翻訳後修飾は発現タンパク質産物に機能や特性を付与するうえで重要な役割を果たし、生物学的プロセスや病態に関与します。例えば、細胞の分化、タンパク質分解、シグナル伝達や調節プロセス、遺伝子発現の制御、タンパク質間相互作用等の多くの細胞内プロセスにおける極めて重要な作用に翻訳後修飾は関与します(1、2、3)。
翻訳後修飾の作用機序
翻訳後修飾は、タンパク質の生合成から分解に至るまであらゆる段階で生じる可能性があります。多くのタンパク質は翻訳完了直後に修飾を受けます。施された翻訳後修飾は、適切なフォールディングの誘導や、新生タンパク質の核や膜等の特定の細胞領域への輸送のために役割を果たします。タンパク質のフォールディングおよび局在化の完了後、さらに別の翻訳後修飾が生じる場合があり、タンパク質の触媒活性/酵素活性等の活性化や不活性化を含む生物学的活性に影響します。また、分解対象のタンパク質に目印をつけるために「タグ」となるタンパク質が共有結合成するような翻訳後修飾もあります。翻訳後のタンパク質は、タンパク質の成熟や活性化の段階的メカニズムを経る際に、切断や官能基の付加を組み合わせた修飾を受けます。
さて、翻訳後修飾はタンパク質のどの部位に生じるのでしょうか?翻訳後修飾は特定のアミノ酸側鎖やペプチド結合箇所で生じ、多くの場合は修飾酵素・脱修飾酵素の作用によって修飾・脱修飾が媒介されます。ヒトプロテオームの5%は翻訳後修飾に働く200種類以上の酵素で構成されることが報告されています。翻訳後修飾に関与する酵素には、アミノ酸側鎖への官能基、タンパク質、脂質、糖鎖の付加あるいは除去を行う酵素、ペプチド結合を切断し特定のアミノ酸配列や調節サブユニットを除去する酵素が含まれます。具体的には、キナーゼ(リン酸化酵素)、ホスファターゼ(脱リン酸化酵素)、トランスフェラーゼ(転移酵素)、リガーゼ、プロテアーゼ等が挙げられます。また、タンパク質自身で自己修飾を行う酵素も数多く存在し、それらは自己リン酸化酵素(autokinase)や自己分解ドメイン(autoprotolytic domain)等の自己触媒性サブユニットを持っています。翻訳後修飾はその特性に応じて、相反する作用を有する酵素同士により可逆的に触媒されるものもあります。例えば、リン酸化修飾反応およびその脱修飾反応が挙げられます。すなわち、キナーゼ(Kinase、リン酸化酵素)の作用によってリン酸基が付加されたタンパク質は、ホスファターゼ(Phosphatase、脱リン酸化酵素)の加水分解作用によってリン酸基が除去されます。この酵素の作用は、リン酸化タンパク質が示す生物活性を逆転させてシグナル伝達を調節します(図1)。
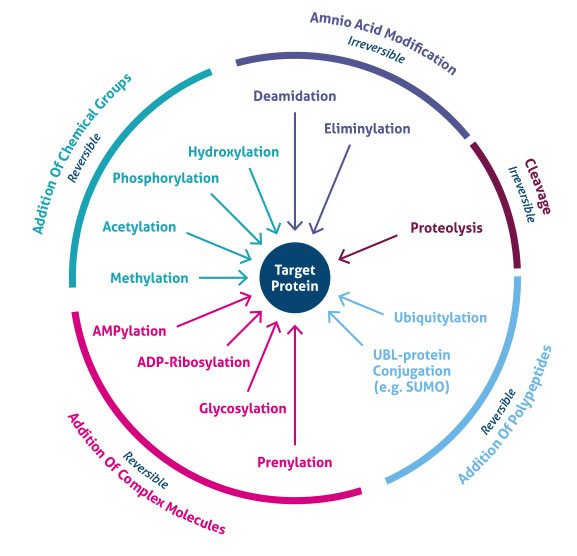
図1. 翻訳後修飾(PTM)の種類
代表的な翻訳後修飾
近年、質量分析(MS:Mass-spectrometry)技術の進歩により、タンパク質の何千種類にも及ぶ翻訳後修飾部位の同定が可能になっています。その結果、高性能な質量分析計と新規手法を組み合わせた測定技術によって、アセチル化、ユビキチン化、O-型グリコシル化、N-型グリコシル化等のいくつかのタイプの修飾が、細胞全域で重要な役割を担っていることが明らかになっています。これまでに小規模な化学修飾(例:リン酸化、アセチル化)から完全なタンパク質の付加(例:ユビキチン化)に至るまで、200種類を超える様々なタイプの翻訳後修飾が判明しています(4、5、6)。
プロテインテックの注目製品:ユビキチン抗体
リン酸化修飾(Phosphorylation)
タンパク質のリン酸化修飾は、最もよく研究対象とされる翻訳後修飾です(図2)。リン酸化修飾はタンパク質機能の調節において極めて重要な役割を果たし、哺乳類のタンパク質の約3分の1はリン酸化修飾を受けていると推定されています。リン酸化は、タンパク質を構成するアミノ酸残基の中で、セリン(Ser、S)、スレオニン(Thr、T)およびチロシン(Tyr、Y)の残基上で生じます。リン酸化は、キナーゼによって触媒され、タンパク質の機能、酵素活性、タンパク質間相互作用、タンパク質局在の制御に影響を与えます。前述の通りリン酸化修飾は可逆的な修飾であり、リン酸化タンパク質はホスファターゼによって脱リン酸化されます。
プロテインテックの関連ブログ:ウェスタンブロットに使用される酵素:適切なツールの選択
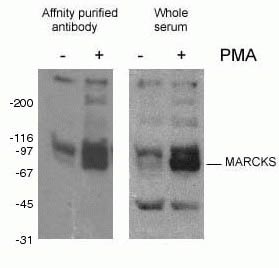
図2. Phospho-Marcksl1(カタログ番号:10018-3-AP、希釈倍率1:1500)を使用したマウスマクロファージ様細胞J774のウェスタンブロットの結果(+:PMA処理、-:未処理)。
グリコシル化修飾(Glycosylation、糖鎖修飾)
グリコシル化とは、共有結合によってポリペプチド鎖に糖が付加することを指します。タンパク質の大部分はリボソームが付着した粗面小胞体でグリコシル化(糖鎖修飾)を受けます。真核生物における最も一般的な糖鎖修飾には、アスパラギン(Asn、N)残基残基の側鎖に糖が結合するN-型糖鎖修飾と、セリン(Ser、S)またはスレオニン(Thr、T)残基の側鎖に糖が結合するO-型糖鎖修飾の2種類が挙げられます。タンパク質の糖鎖修飾を実験的に示す場合、グリコシダーゼ処理を施したサンプルを用いてウェスタンブロットを実施することにより、目的タンパク質に糖鎖修飾が存在するのか否かを確認することができます。
プロテインテックの関連ブログ:ウェスタンブロットに使用される酵素:適切なツールの選択
ユビキチン化(Ubiquitination)
タンパク質のユビキチン化とは、タンパク質のリシン(Lys、K)、システイン(Cys、C)、セリン(Ser、S)、スレオニン(Thr、T)の残基上、またはタンパク質のN末端に直接ユビキチン分子が共有結合することを指します。ユビキチン分子は、76残基のアミノ酸からなる分子量の小さなタンパク質(8.6 kDa前後)であり、ほぼすべての種類の組織に発現しています(図3)。ユビキチン化は3種類の酵素(E1、E2、E3)に触媒される連続的な酵素反応によって生じます。一連の酵素は、基質タンパク質(ユビキチン)の選択、基質タンパク質の活性化、修飾対象タンパク質との結合、ユビキチンの連結反応を担います。タンパク質はモノユビキチン化(1分子のユビキチンが付加)される場合もあれば、ポリユビキチン化(多数のユビキチンが付加)される場合もあります。ポリユビキチン化される場合は、最初に付加したユビキチン分子にさらにユビキチン分子が鎖状に付加します。ユビキチン修飾は、プロテアソームを介したタンパク質分解のための目印になります。また、ユビキチン化は、細胞内のシグナル伝達、膜タンパク質の内在化、転写の開始や制御においても重要な役割を果たします。
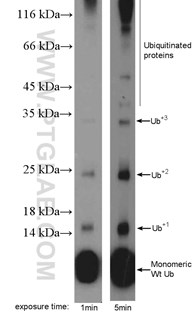
図 3. MDA-MB-453細胞をSDS-PAGE後、ユビキチン抗体(カタログ番号:10201-2-AP、希釈倍率1:600)を使用して実施したウェスタンブロット。
翻訳後修飾が正常な機能や疾患に与える影響
特定のタンパク質とその翻訳後修飾の解析は、心疾患、がん、神経変性疾患、糖尿病等で重要な研究対象となっています(7)。翻訳後修飾を介したタンパク質制御に関わる研究を遂行する場合、特異的な検出方法や精製方法等の選択や開発が課題となっていました。しかし現在までに、このような技術的課題は様々な新規のプロテオミクス技術の開発によって解消されつつあります。
- G Grotenbreg, H Ploegh. Chemical biology: dressed-up proteins. Nature. 2007 Apr 26;446(7139):993-5.Chemical biology: dressed-up proteins.
- R Geiss-Friedlander, F Melchior. Concepts in sumoylation: a decade on. Nat Rev Mol Cell Biol. 2007 Dec;8(12):947-56.
- R S Morrison, et al. Proteomic analysis in the neurosciences. Mol Cell Proteomics. 2002 Aug;1(8):553-60.
- G Duan, D Walther. The roles of post-translational modifications in the context of protein interaction networks. PLoS Comput Biol. 2015 Feb 18;11(2):e1004049.
- P Minguez, P Bork, et al. Deciphering a global network of functionally associated post-translational modifications. Mol Syst Biol. 2012 Jul 17:8:599.
- P Minguez, I Letunic, P Bork, et al. PTMcode: a database of known and predicted functional associations between post-translational modifications in proteins.Nucleic Acids Res. 2013 Jan;41(Database issue):D306-11.
- B György, et al. Citrullination: a posttranslational modification in health and disease. Int J Biochem Cell Biol. 2006;38(10):1662-77.



